摘要: 幾年前,連研究人員都不願使用DNA來存儲數據,覺得這麼做太超前了,不具有任何實用價值。今天,你可以使用合適的軟件和生化模塊來擴展的PostgreSQL,並在DNA上運行SQL 。
幾年前,連研究人員都不願使用DNA來存儲數據,覺得這麼做太超前了,不具有任何實用價值。今天,你可以使用合適的軟件和生化模塊來擴展的PostgreSQL,並在DNA上運行SQL 。
當下全世界的數據浪潮來勢兇猛,不僅超出了我們理解數字和衡量單位(比如澤字節)的能力,還超出了我們存儲海量數據的能力。
一切都變得數字化,一切都日益在基於算法的應用軟件上運行,這些算法拿數據來訓練,反過來生成更多的數據,饋送給為更多的下游應用軟件和算法......結果可想而知。
簡而言之,按照這種步伐,很快就沒有足夠的數據存儲和計算材料以滿足需求。這就是為什麼人們現在一直在尋找替代的存儲介質以存儲數據。使用DNA存儲數據乍一聽很奇怪,實際上大有意義。現在研究人員已取得了重大突破,他們因而能夠將DNA存儲整合到PostgreSQL的這種流行的開源數據庫中。
DNA是一種信息編碼機制
究其核心,DNA是一個數據存儲層。DNA由四種基礎部分組成:腺嘌呤,鳥嘌呤,胞嘧啶和胸腺嘧啶(又名AGCT)。DNA由這四個鹼基組成三個核苷酸形成的三聯體(名為密碼子)。密碼子是給人體細胞下達蛋白質形成指令的單位。
我們的信息技術基礎設施基於以比特(包括兩個數字:0和1)。來存儲信息,而DNA信息存儲在四個潛在鹼基單位的串中為了將非遺傳信息存儲在DNA中,我們必須先將二進制數據從比特轉換成DNA數據的四單位(AGCT)結構。
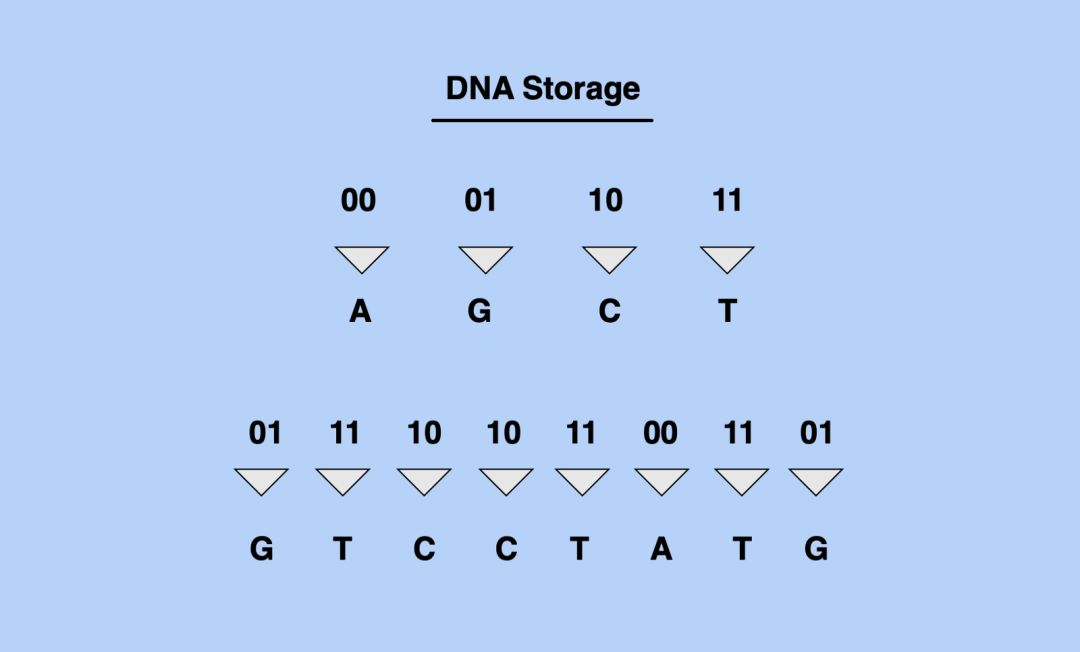
▲將比特轉換成DNA序列
理論部分實際上相當簡單。與使用矽或磁性介質(其工作原理基於將狀態存儲為1和0序列的能力)一樣,我們可以使用DNA,存儲A,G,C和Ť組成的序列。但是這實際上如何運作 - ?該如何將數據寫入到DNA和從DNA中讀取數據?
這可能聽起來太過遙遠,但分子技術的進步使其變得切實可行,儘管並不稱心如意。這一切意味著,確實能夠以一種可以在DNA上存儲和檢索信息的方式來編碼信息,分別利用DNA合成和DNA測序。
比如說,微軟已展示了世界上第一個自動化的DNA數據存儲和檢索系統你可能想知道這個DNA來自哪裡,告訴你:這是合成的DNA,生成合成DNA的陣列是系統的一部分。
天然存在的DNA呈現有兩條核苷酸鏈的雙螺旋這種結構。相比之下,用於數據存儲的DNA是單鏈核苷酸序列,又叫寡核苷酸(低聚),它是使用每次一個核苷酸來組裝DNA的化學過程合成的。
法國通信系統工程師學校與研究中心(通信系統工程師學校與研究中心)數據科學系助理教授Appuswamy和倫敦帝國理工學院SCALE實驗室負責人Heinis最近發表了DNA存儲方面的開創性成果。
使用DNA在現實世界存儲數據
Heinis和Appuswamy在創新數據系統研究大會上發表了題為“OligoArchive:在DBMS存儲層次結構中使用DNA”的研究論文

雖然他們並非最先使用DNA來存儲和檢索數據,卻最先針對結構化數據這麼做,與現成數據庫集成起來,而且不僅限於存儲,還實現了計算。
DNA作為數據存儲層方面要認識到的第一點是,每次執行寫入操作時,都必須合成寡核苷酸。這實際上將如何做到?實驗室技術人員是否要待命執行此操作,並為用於化學過程的原材料“重新灌滿油箱”?
據Appuswamy和Heinis聲稱,並非如此,微軟用自動化的DNA存儲和檢索系統演示了其在這方面的價值。結果表明,可以在無需人參與的情況下操作這種陣列。就像沒人監管數據中心的日常運營一樣(維護除外),基於DNA的數據中心將同樣如此。
不過,我們離合成DNA陣列替換傳統硬盤還遠得很。首先,以這種方式存儲數據的現代技術速度非常慢。最初,存儲1兆字節的數據需要科學家花一周時間。
Appuswamy和Heinis都認為這方面需要做更多的工作。雖然這超出了他們自身研究的範圍,所以只好等生化組合過程趕上來,但他們確實讓人看到了希望。
首先,他們特別指出存儲速度在變得越來越快,目前每秒可以存儲數KB。比如說,儘管與SSD相比速度仍然慢得要命,但已是相當大的進步。這個速度對於Appuswamy和Heinis的研究設想的使用場景:歸檔存儲而言實際上可以接受。
數據庫引擎使用三層存儲層次結構,這種層次結構包括價格/性能特點大不一樣的眾多設備。性能層存儲高性能OLTP和實時分析這類應用訪問的數據。
容量層存儲對延遲不敏感的批處理分析這類應用訪問的數據。歸檔層用於存儲極少訪問的數據,比如在安全合規檢查或法務審計期間。如今,磁帶通常用於這一層。
OligoArchive改變了數據庫存儲層次結構:它將基於磁帶的歸檔層換成了基於DNA的歸檔層合成DNA使用額外的預防措施來加以存儲;至於將基於DNA的存儲用於普通設備效果有多好還成問題。但數據和數據庫進入雲端是大勢所趨,只要你的數據安全地存儲在數據中心,它在最終用戶眼裡就是黑盒子。
在DNA上運行SQL
Appuswamy和Heinis還特別指出,儘管速度仍很慢,但DNA存儲在並行處理方面大有潛力這是由於DNA存儲數量充足,成本低廉。 - 或者更準確地說,希望最終會如此,按目前情況來看,存儲1分鐘的高質量立體聲將花費10萬美元。
雖然使用合成DNA用於大規模存儲仍然成本過於高昂,但Appuswamy和Heinis表示,他們預計每一次科技突破(包括存儲技術)通常會使成本大幅下降。
如果合成寡核苷酸在經濟上變得可行,讓許多寡核苷酸滿足存儲需求自在情理之中。這意味著讓許多DNA存儲單位並行操作這方面巨大潛力。雖然並非每種算法的每個方面都可並行化,但對於果真可並行化的算法而言,可以大幅提升速度。這引出了一個關鍵點。
就在不久前,DNA還被用於存儲非結構化文件,無論是文本,視頻或諸如此類的數據。Appuswamy和Heinis所做的是將DNA存儲集成到關係數據庫中。他們拿來標準數據庫基準測試TPC -H中包含的數據和查詢,在PostgreSQL的實例上運行TPC-H。不是串行訪問,而是隨意選擇數據。
將結構化數據存儲在數據庫系統中,後端使用DNA,並通過SQL來查詢,這在今天已成為現實。
研究人員為PostgreSQL的構建了歸檔和恢復工具(pg_oligo_dump和pg_oligo_restore),這些工具在DNA上對關係數據執行感知模式的編碼和解碼,然後他們用這些工具將12KB TPC-H數據庫歸檔到DNA,執行體外計算,然後再恢復該數據庫。
這意義重大這意味著現在DNA存儲還可以支持SQL操作,選擇性地訪問和處理部分數據請注意:。數據並不被提取到數據庫以便在那裡執行操作.Appuswamy和Heinis找到了一種方法在寡核苷酸中處理SQL連接之類的操作。這超出了生化存儲的範疇,還涉及生化計算。
用於編碼和解碼進出DNA的信息的技術存在著缺陷;然而要做到這一點,研究人員就得處理與這些缺陷有關的一堆問題在DNA上執行操作需要專門的編碼技術,這些技術可以生成適合生化操作的寡核苷酸讀取DNA數據目前很容易出錯,而以前的研究依賴數據過度表示:數據以多個副本寫入,因此即使原始數據被破壞,還有備份。
相比之下,Appuswamy和Heinis依賴元數據。他們在編寫的代碼段中添加了一些額外的數據,利用數據庫模式感知功能。他們表明,這可以在編碼(寫入)過程中提高密度,並有助於在解碼(讀取)過程中識別錯誤他們特別指出實際效果比預期的好 - 一點元數據就大有幫助。
DNA是數據的未來嗎?
儘管技術堆棧的某些部分還不成熟,但這是一項重大突破。讓已有的數據中心擁有充足的存儲資源可以改變遊戲規則。但將DNA這種數量充足的材料充當存儲和計算的可行介質具有重大影響,遠非我們所能想像。
這可能只是朝這個方向邁出的第一步,但每段旅程都從第一步開始.Appuswamy和Heinis並非單槍匹馬開展這項工作,他們也不會憑一己之力開展進一步的研究。他們的項目OLIGOARCHIVE一直在取得進展,這歸功於與法國蔚藍海岸大學(UCA)和CNRS的其他研究人員進行合作,因而得以壯大研究團隊,並擴大研究範圍。
通信系統工程師學校與研究中心,CNRS,ICL,UCA以及DNA合成初創公司Helixworks已獲得歐盟資助,以進一步開展DNA存儲方面的研究。該系統將旨在支持編碼數據,將數據合成為DNA,並通過測序讀回數據這整個過程完全實現自動化。它將存儲眾多不同類型的數據,並實現近數據處理和數據的較精確檢索。

將數據存儲在DNA中方面的進一步研究將得到歐盟的資助。
該項目通過未來和新興技術(FET)歐盟計劃獲得資金,該計劃投資致力於研究全新未來技術方面新想法的早期階段項目,在早期階段很少有研究人員開展項目課題。
Appuswamy和Heinis提到,到目前為止,感興趣的主要是其他研究人員,不過微軟是個例外。倒不是這方面有任何實際成果,而是眼下微軟似乎比其他任何公司抱有更大的興趣。
在這項技術方面獲得優勢可能意味著主導未來,因為這個領域的突破將帶來巨大影響Appuswamy和Heinis特別指出,人們的態度表明了這一點:“幾年前,人們會覺得這遙不可及。而今天,我們告訴他們我們在做的工作後,他們的態度是'告訴我們更多'“。
論文全文:







轉貼自: 鍊數成金
若喜歡本文,請關注我們的臉書 Please Like our Facebook Page: Big Data In Finance


留下你的回應
以訪客張貼回應